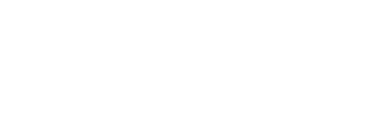A interação entre os fármacos e o cérebro
POR ANA PAULA ACAUANOs receptores de canabinoide no cérebro, os mesmos que reconhecem substâncias psicoativas derivadas da maconha, ao interagirem com medicamentos, podem trazer efeitos benéficos à saúde. Estudo publicado na revista científica Current Medicinal Chemistry, da Bentham Science Publishers, dos Emirados Árabes Unidos, com sede em vários países, avança nessa investigação ao integrar simulações computacionais. “Encontramos uma abordagem que consegue prever o encaixe da chave na fechadura, ou seja, dos possíveis fármacos com a proteína – o receptor. Isso de forma precisa, porque comparamos com informações experimentais, chegando a resultados próximos”, destaca o professor Walter de Azevedo Jr., da Escola de Ciências da Saúde e da Vida, orientador da tese de doutorado sobre o tema, da médica anestesista Silvana Russo, no Programa de Pós-Graduação em Biologia Celular e Molecular.
Um alvo muito estudado para entender a relação com o sistema nervoso central ainda não tinha sido investigado computacionalmente. O trabalho obteve destaque mesmo com a pouca disponibilidade de dados biológicos sobre o receptor de canabinoide. Se houvesse um volume maior de informações, a confiabilidade aumentaria.
Um dos desafios, na visão de Silvana, é desmitificar o tema, visto com preconceito pelo uso da maconha. “Temos de mostrar que é um assunto sério e que há medicamentos importantes relacionados com esse receptor.” Já se sabe seu papel no tratamento da epilepsia e é estudado seu efeito na dor. Substâncias utilizadas para obesos alcançaram boa resposta, mas em alguns pacientes causaram depressão, sendo retiradas do mercado. O estudo de bioinformática incluiu esses fármacos – Taranabant e Otenabant. “Mostramos a interação entre as moléculas da proteína e do ligante – das drogas, o que pode servir de base a outros laboratórios na investigação de modificações nos fármacos que não gerem o efeito colateral”, explica Silvana.
Imagem divulgada na capa da Current Medicinal Chemistry mostra o padrão que serve de base para as interações do receptor canabinoide (representado pela cor roxa) com os fármacos. O azul representa as ligações envolvendo carbono; o vermelho, oxigênio; e o branco, hidrogênio
Essas ferramentas computacionais não queimam etapas. É preciso fazer testes in vitro das células e com animais, mas a bioinformática simplifica o processo. “Cortamos um caminho. Em vez de analisarem um milhão de substâncias, a gente sugere algumas que têm mais potencial, poupando tempo e dinheiro”, complementa Silvana.
Agora o grupo vai investigar a dipirona, uma medicação muito usada como analgésico e antitérmico. É uma das apostas para ativar o receptor de canabinoide. “Simulações computacionais mostram que pode ser interessante para outros usos ou mesmo gerar estudos para esclarecer seu real efeito no organismo.” Artigo sobre o tema também foi aceito para publicação na Current Medicinal Chemistry.
Referência em bioinformática |
Bolsista de Produtividade em Pesquisa do CNPq nível 1D (o máximo é 1A), o professor Walter Filgueira de Azevedo Jr. atua na PUCRS desde 2005. Com graduação, mestrado e doutorado em Física, todos pela USP, é livre docente pela Universidade Estadual Paulista Júlio de Mesquita Filho (2004) em Biofísica Molecular e pós-doutor pela instituição (1997-1998), na mesma área e em Química de Macromoléculas. Reconhecido na área de docagem molecular, investigando o ajuste entre as proteínas e os ligantes – possíveis fármacos –, o professor tem 4.519 citações, de 155 trabalhos, no portal de periódicos Web of Science (fator de impacto H: 6, ou seja, no mínimo, 36 artigos publicados e esses receberam pelo menos 36 citações cada). Na Scopus, chega a 4.725 citações de 161 artigos e, em outros indexadores, (Google Acadêmico), 6.054 citações de 158 artigos. É membro do corpo editorial dos periódicos Current Medicinal Chemistry – do qual é editor da seção de Bioinformática em Desenho e Descoberta de Fármacos –, Current Bioinformatics, PEERJ, Current Drug Targets e Methods in Molecular Biology (Springer Nature). |
Busca de novos conhecimentos |
A médica anestesista e intensivista Silvana Russo é formada pela Universidade Federal de Pelotas (1989). Fez toda a carreira profissional para depois optar pela pós-graduação. É mestre em Medicina e Ciências da Saúde pela PUCRS, orientada pelo professor André Palmini. Ao cursar a especialização em Bioinformática em 2018, se desafiou a buscar novos conhecimentos e acabou no Doutorado em Biologia Celular e Molecular. “A maioria das pessoas prefere ficar no campo em que domina. Eu gosto de aprender coisas novas.” Interessada em medicina da dor, viu a notícia de que um grupo havia decifrado a estrutura do receptor canabinoide. Sua tese aborda essa proteína e os desafios no desenho de fármacos com enfoque nas ferramentas da bioinformática. |

O mestre e os discípulos: Walter (E), Amauri e Gabriela
Software ajuda na construção de modelos
No trabalho de conclusão do curso de Especialização em Bioinformática da PUCRS, Amauri Duarte da Silva, orientado pelo professor Walter de Azevedo Jr., criou um software para auxiliar no desenvolvimento de novos fármacos, utilizando aprendizado de máquina e uma técnica que ajuda a prever a afinidade entre um ligante (um possível medicamento) e uma proteína. De nome Taba – Tool to Analyze the Binding Affinity – é uma ferramenta de código aberto, disponível para a comunidade científica. Os resultados foram publicados na capa do Journal of Computational Chemistry, sendo superiores aos obtidos por outros programas.
Para estudar a interação de fármacos com proteínas, pode ser utilizada a Física Quântica, mas o custo computacional é muito alto. “Com a simplificação, a conta fica mais rápida, só que pouco realista. Temos que achar um meio termo”, esclarece o professor. Hoje é feita uma equação para cada proteína.
A ferramenta calcula as distâncias médias entre pares de átomos envolvendo proteína e ligante. Através de valores de afinidade experimentais, pode-se prever o que ocorre com outras substâncias estudadas. Além da ativação da proteína, os fármacos também são utilizados como inibidores de receptores relacionados a alguma doença.
Graduado em Administração de Empresas – Análise de Sistemas pela PUCRS, Duarte atua no Serviço Federal de Processamento de Dados. Começará o mestrado em Biologia Celular e Molecular em 2020. A estudante de iniciação científica Gabriela Bitencourt-Ferreira participou do estudo, realizando testes adicionais da ferramenta.
Informações sobre a ferramenta e as pesquisas:
https://azevedolab.net